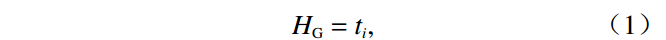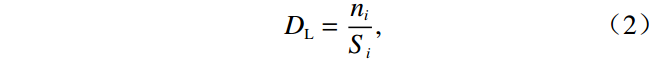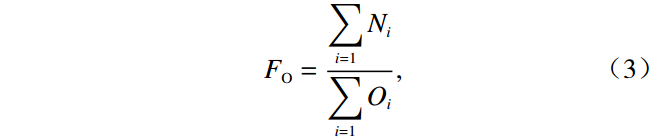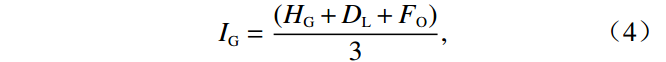Effects of forest grazing on soil properties and microbial community in the Northeast China Tiger and Leopard National Park
-
摘要: 选取典型的放牧区和非放牧区,采用分层抽样设立了100个样方(放牧区47个、非放牧区53个),采用高通量测序数据并结合大数据分析的方法探讨了森林放牧对土壤理化性质和土壤微生物群落组成的影响.研究结果显示:放牧显著增加了土壤密度(P = 0.005)和电导率(P = 0.045),降低了土壤含水量(P = 0.022),表明家畜的活动可能已经破坏土壤结构和降低土壤保水能力;放牧区的土壤可交换K+(P = 0.015)与Ca2+(P = 0.006)、全钙质量分数 (P = 0.004)显著高于非放牧区,表明放牧干扰加速了国家公园土壤养分积累.通过高通量测序技术,依据97%相似性原则,将细菌序列划分为9488个可操作分类单位(operational taxonomic units,OTUs),真菌序列划分为5676个OTUs.放牧区与非放牧区的土壤细菌和真菌群落组成差异显著,坡度、土壤电导率和植物丰富度是影响土壤细菌群落组成的主要因素,而真菌群落组成受到放牧强度、海拔、坡度、植物丰富度、土壤电导率、全氮和速效磷的显著影响.进一步分析表明:变形菌门的相对丰度与放牧强度、海拔、电导率显著正相关,而与植物丰富度显著负相关;放线菌门的相对丰度与电导率显著正相关;绿弯菌门的相对丰度与电导率、植物丰富度显著正相关;担子菌门的相对丰度与海拔、坡度显著正相关,而与电导率、速效磷质量分数、植物丰富度显著负相关;子囊菌门和孢霉菌门的相对丰度与电导率、速效磷质量分数、植物丰富度显著正相关,而与海拔、坡度显著负相关;罗兹菌门的相对丰度与全氮、速效磷质量分数、植物丰富度显著正相关,而与海拔、坡度显著负相关.本研究表明,长期林下放牧对东北虎豹国家公园土壤理化性质和土壤微生物群落结构产生了不同影响,考虑到土壤与其微生物之间的强相互作用,可以预计土壤微生物群落结构的变化将极大地改变土壤功能,这对指导野生动物栖息地的恢复具有重要意义.Abstract: Long-term forest grazing is a major anthropogenic disturbance in Northeast China Tiger and Leopard National Park, posing great threat to forest ecosystems due to heterogeneous and unpredictable changes in forest structure, function, and biodiversity. Understanding the responses of soil properties and microbial to grazing is useful for forest management and biodiversity conservation. The effects of long-term forest grazing on soil physicochemical properties and soil microbial community composition in the Northeast China Tiger and Leopard National Park were assessed in this study. Soil samples were collected in the summer in 2021, from 47 cattle-grazed forest plots and 53 un-grazed forest plots. High-throughput sequencing technique was used to analyze soil bacterial and fungal communities in two forest areas. Generalized linear mixed model and non-metric multidimensional scaling were used to assess effects of forest grazing on soil properties and microbiome. Continuous forest grazing was found to significantly increase soil bulk density (P = 0.005), soil conductivity (P = 0.045), but decrease soil water content (P = 0.022); livestock grazing may therefore damage soil structure and reduce soil water retention capacity. Soil exchangeable K+ (P = 0.015) and Ca2+ (P = 0.006), soil total calcium (P = 0.004) were significantly higher in grazed forests than un-grazed forests, grazing disturbance therefore accelerated soil nutrient accumulation in the National Park. A total of 9488 bacterial operational taxonomic units (OTUs) and 5676 fungal OTUs were obtained according to a 97% sequence similarity level. Non-metric multidimensional scaling revealed that composition of soil bacterial and fungal community differed significantly between grazed and un-grazed forests. Soil bacterial community composition was primarily influenced by slope, electrical conductivity, plant richness, whereas fungal community composition was significantly influenced by grazing intensity, elevation, slope, plant richness, electrical conductivity, total nitrogen and available phosphorus. The relative abundance of Proteobacteria was found significantly positively correlated with grazing intensity, elevation and electrical conductivity, but negatively correlated with plant richness. Relative abundance of Actinobacteriota was positively correlated with the electrical conductivity. Relative abundance of Chloroflexi was significantly correlated with electrical conductivity and plant richness. Relative abundance of Basidiomycota was significantly positively correlated with elevation and slope, but negatively correlated with electrical conductivity, available phosphorus and plant richness. Relative abundance of Ascomycota and Mortierellomycota were found significantly positively correlated with electrical conductivity, available phosphorus and plant richness, but negatively correlated with elevation and slope. Relative abundance of Rozellomycota was significantly positively correlated with total nitrogen, available phosphorus and plant richness, but negatively correlated with elevation and slope. These data suggest that forest grazing practices affect soil properties and microbes differently. Given the strong interaction between soil and microbiome, changes in soil microbial community structure can be expected to substantially alter soil function. This will have important ecological service implications, particularly for wildlife habitat and carbon storage in key regional priorities for biodiversity.
-
0 引言
随着人口增长和粮食需求的增加,陆地上大量自然植被变成耕地或牧场,导致全球物种多样性快速丧失[1−2].放牧作为全球最广泛的土地利用类型之一,是影响土壤特征、动植物多样性和生态系统功能的重要生物过程[3−4].例如:过度放牧会引起土地利用类型的变化,降低植被覆盖度,导致野生动物栖息地破碎化,最终引起生物多样性丧失以及生态系统功能的降低;放牧还会造成物种入侵[5],取代已灭绝的本土食草动物的生态功能[6]和改变物种的生态性状[7];依据中度干扰假说理论,放牧对于维持植物群落物种多样性也有一定的贡献[8].目前,家畜放牧已占全球约1/4的陆地面积[9],甚至出现在保护区、国家公园或其他自然保护地,并在森林生态系统中加速扩张[10].持续的放牧对自然资源和生态环境产生了巨大的压力,因此有必要更好地了解放牧对生态系统的综合影响,以实现生态保护目标.
过度放牧导致的土壤退化已经成为一个全球性问题,越来越受到生态学家的关注[11−12].土壤的性质是用于评价土壤健康的重要指标,对于维持生态系统功能和预防土地退化至关重要[11,13].放牧伴随的牲畜践踏和排泄物分解等能直接或间接地改变土壤结构和养分积累.家畜的践踏行为能够降低土壤孔隙度[14]、增加土壤密度[15],导致空气和水的渗透性下降[16];粪便和尿液的沉积能促进养分循环,增强土壤净氮矿化和氮硝化速率,提高土壤养分有效性[17−18],降低土壤pH,加速土壤氮循环[12].此外,放牧引发的土壤结构和养分循环变化可以显著影响土壤微生物群落的组成及其功能[11,19].
土壤微生物是地球生物多样性的重要组成部分,在陆地生态系统中扮演着重要角色[20],在调节有机质分解及养分矿化等方面发挥着关键作用.由于高通量测序技术的不断发展,对土壤微生物群落组成和功能的认知正在增加[21].土壤微生物对环境因子,如土壤类型、pH、植被等十分敏感,可以对土壤性质和功能的变化起到良好的指示作用[22−23].在森林生态系统中,放牧干扰能够改变土壤微生物群落的多样性和组成,显著影响土壤碳、氮的有效性以及土壤呼吸[18,24],进而改变土壤的功能及养分供给模式[25];与此同时,放牧也能够通过改变土壤理化性质来调控土壤微生物的群落组成[26],如在澳大利亚东部地区的森林生态系统中,放牧强度的增加显著降低了土壤碳含量,进而抑制了优势细菌放线菌门(Actinobacteriota),增加了优势真菌子囊菌门(Ascomycota) [27].
在森林中,家畜的活动能占据野生动物栖息地,代替野生大型食草动物,它们的存在不仅对野生动物和植物产生直接影响,还会通过营养级联效应改变土壤结构和功能[3−, 4].家畜的啃食行为能够改变植物生产力和群落组成,进而间接影响土壤微生物群落[28].放牧引起的植被消失会导致地下植物生物量的偏向分配[29],增加根系分泌物,如酶、有机物和多糖[30],最终提高土壤中病原体丰度.此外,高强度的放牧干扰会使得速生植物物种产生高营养的凋落物,有利于土壤细菌生长,而低放牧强度则加速了低营养凋落物的积累,有利于土壤真菌生长[31].
东北虎豹国家公园有超过30年的放牧历史,存在大面积的牧场与高强度的家畜活动[32-33],每年的4—11月,家牛被放养到森林中,处于自由放牧状态,无人看管[34].长期的放牧干扰已经使当地野生动物栖息地面临退化,阻碍了野生动物种群扩散[33,35],导致灌草层生物量下降超过20%[36].根据营养级联假说,放牧不仅对地上部食草动物、植物产生了影响,家牛的活动还会引发下行效应对地下部土壤产生一定的影响[11−12].因此,本研究在东北虎豹国家公园采用分层抽样的方法选取典型的放牧与非放牧样方,对样地内植物群落进行全面调查,并收集样地的环境和土壤数据;利用高通量测序技术,探究森林放牧对土壤理化性质及土壤微生物群落组成的影响,并回答以下3个问题:1) 林下放牧是否改变了国家公园土壤理化性质?2)放牧强度的增加,森林土壤理化性质如何变化?3) 放牧干扰是否为国家公园土壤微生物群落结构的主要影响因素?
1 材料与方法
1.1 研究区概况
研究区域位于东北虎豹国家公园东部(129°05′00″~131°18′48″ E, 42°31′06″~ 44°14′49″ N),东与俄罗斯接壤,南与朝鲜隔江相邻.该区域海拔范围5~1477 m,气候类型为温带大陆性季风气候,年均气温为5.60 ℃,年平均降水量618 mm,无霜期为110~160 d,土壤为暗棕壤.全年季节分明,其中5—10月为生长季,11—翌年4月为非生长季.森林覆盖率 > 92%,森林类型以次生落叶阔叶林为主[37].长期红外相机监测数据表明,至少存在28种野生兽类[38],以东北虎(Panthera tigris altaica)和东北豹(P. pardus orientalis)为旗舰兽类.研究区拥有长期的放牧历史,家牛的密度达到8~12只·km−2[34].
1.2 牧场调查与样地设置
2015年夏季对国家公园东部核心区的牧场分布进行了调查,调查面积为4000 km2.调查对象为所有经营牧场,通过查阅经营日志获取放牧基础数据,包括家畜类型、家畜数量、成立时间;并使用便携式GPS(HOLUX Inc., m-241, 中国台湾)测量牧场范围,利用ArcGIS 10.3(ESRI Inc., Redlands, CA, USA)计算牧场面积.根据牧场调查的结果,将研究区在景观尺度上划分为了典型的放牧区和非放牧区.
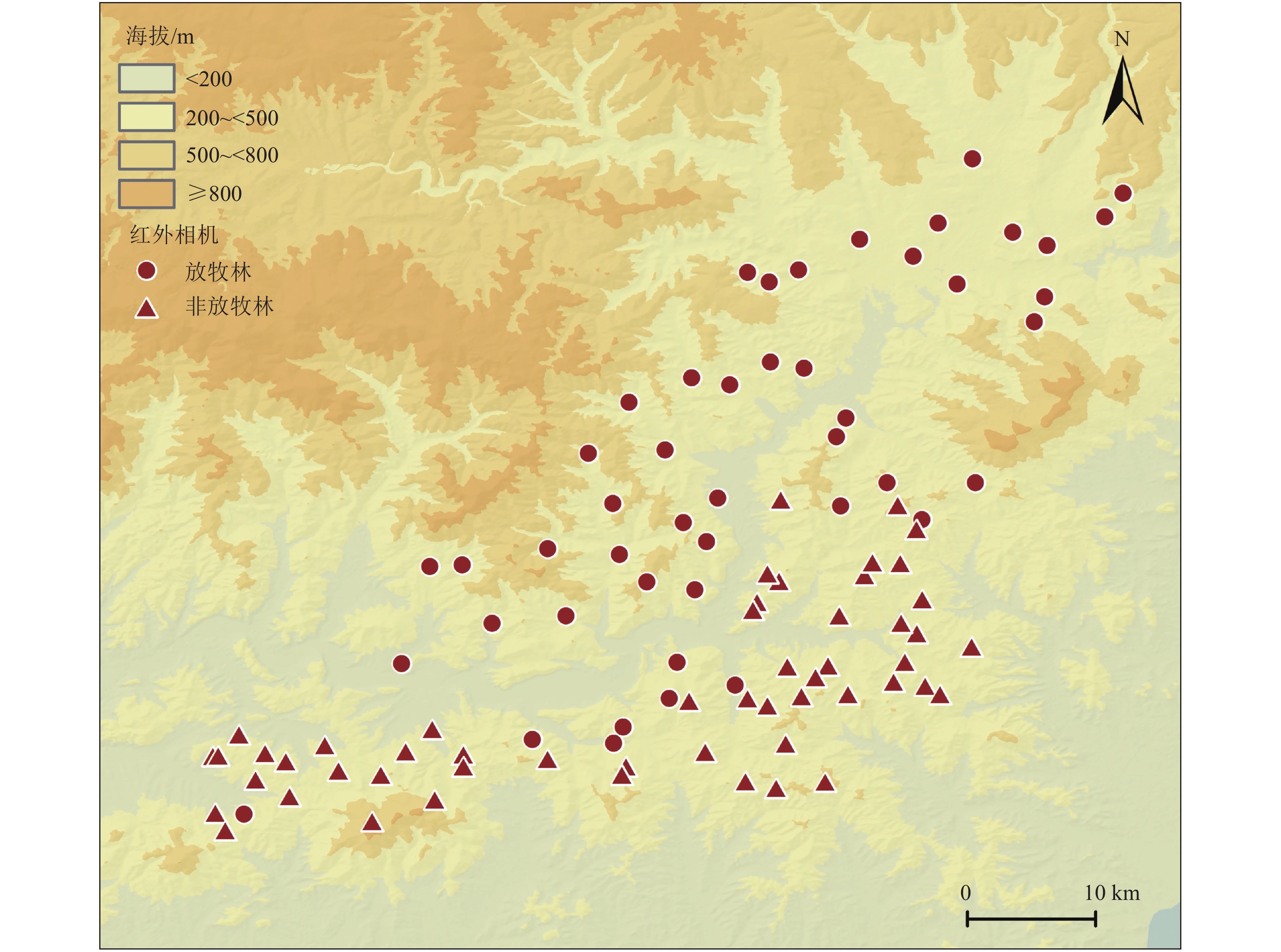
为消除地形因子及人为主观选取采样点造成的误差,本研究采取环境因子辅助采样方法设置样地[39].选取的环境因子包括海拔、坡度、坡向、沿等高线曲率、沿剖面曲率、表面凹凸度和林型,除林型外其他环境因子根据分辨率为25 m的数字高程模型(digital elevation model)计算获得.我们将研究区划分为100 m × 100 m的栅格,采用K-means算法对所有环境因子进行聚类分析[40],并对分类结果进行方差分析以验证各组间地形差异是否显著.聚类结果显示,研究区可分为4类地形(附件1),每类地形重复取样点见附件2.采用典型样地法,在每类地形内依据放牧与否进行随机分层抽样,共设置100个样地,其中放牧区47个,非放牧区53个(图1).
1.3 样地调查
使用GPS(UniStrong,G138BD,China)和罗盘(SUUNTO,MC.2,Finland)对样地定位,并记录海拔和坡度.每个样地设立20 m × 20 m样方,对样方内乔木胸径(DBH)≥3 cm进行测量,记录其DBH和树种等数据,每个样地内随机选取3 个5 m × 5 m 的灌木样方,5个1 m × 1 m 的草本样方,记录所有出现的物种,测量其高度与盖度.每个样地架设1台红外相机(相机的详细设置见文献[38]),对样地内家畜与野生动物种群与活动进行监测.
1.4 样品采集与土壤理化性质测定
为防止干旱、降雨等天气因素的影响,所有土壤样品均选择在匀质天气进行采集.每个样地在东北至西南方向对角线设立等距的3个重复采样点.采用便携式土壤三参数测定仪(Delta-T,WET-2,UK)测定土壤电导率,采用环刀法计算土壤密度.在每个采样点取10 cm × 10 cm × 20 cm土芯,混合后采用四分法取200 g,过2 mm筛,每个样品分为2份:1份样品(约50 g)装于50 mL无菌PE管中,用于土壤总DNA的提取;另1份样品(约150 g)装入自封袋中,用于测试土壤理化性质.所有样品置于低温冰箱中暂存,运回实验室.50 g放置于−80 ℃冰箱保存,用于土壤总DNA的提取,150 g在室内进行风干后将样品装袋密封保存.本研究测定的土壤化学性质指标包括pH,有机碳质量分数,含水量,全元素(氮、磷、钾、钙、镁、钠)、速效磷、速效钾、交换性离子(钾、钙、镁、钠)质量分数,所有指标均按照标准流程测定[41].
1.5 土壤总DNA的提取与高通量测序
选取20个典型的放牧样方与20个非放牧样方的土壤样品用于高通量测序.每个土壤样品取0.35 g,利用E.Z.N.A.® soil DNA kit试剂盒(Omega Bio-tek, Norcross, GA, U.S.) 提取土壤总DNA.将得到的DNA利用NanoDrop 2000 (Thermo Scientific,USA) 和琼脂糖凝胶电泳仪检测样品的浓度和完整性(琼脂糖凝胶质量分数为1%,电场强度5V·cm−1和电泳时间20 min).PCR:选用338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R (5'-GGACTACHVGGGTWTCTAAT-3') 扩增细菌16S rDNA V3-V4高变区;利用ITS1(5'-CTTGGTCATTTAGAGGAAGTAA-3') 和 ITS2(5'-GCTGCGTTCTTCATCGATGC-3')扩增真菌ITS1or ITS2.利用2%琼脂糖凝胶检测PCR扩增产物,以确定扩增的成功率.使用TruSeq® DNA PCR-Free Sample Preparation Kit进行文库构建.测序工作由上海美吉生物医药科技有限公司完成.基于Illumina 公司的Miseq PE300测序平台,利用单端测序的方法,构建小片段文库进行单端测序.
1.6 高通量测序数据的处理
测序完成后,使用FASTP (https://github.com/OpenGene/fastp,version 0.19.6) 软件对双端原始测序序列进行质控[42];使用FLASH (http://www.cbcb.umd.edu/software/flash,version 1.2.11) 软件进行拼接[43];使用UPARSE软件[44] (http://drive5.com/uparse/,version 7.1)在97%相似性水平下将序列聚类成为OTUs (operational taxonomic units),注释采用细菌SILVA数据库(Silva 138.1)和ITS的真菌UNITE数据库(Unite 8.0)通过feature-classifier插件进行物种注释,后续分析的OTUs数据均是按照样品最小序列数对OTU表进行抽平处理.
1.7 放牧强度
放牧强度(grazing intensity,IG)通常定义为家畜密度,但家畜密度仅反映了家畜数量,并未考虑长期放牧的累加效应和家畜行为活动的影响,并不能客观反映放牧干扰的影响[45-46].因此,本研究考虑每个样地的放牧历史、家畜密度和出现频度,按照三者权重计算放牧强度.其中:放牧历史(grazing history,HG)定义为牧场设立年限;家畜密度(livestock density,DL)定义为每平方千米内家畜数量;家畜出现频度(frequency occurrence,FO)根据放牧期(2019年7—10月)红外相机观测数据[38]计算,每3 d作为一个调查周期,统计家畜在每个周期内的出现情况,出现记为1,不出现记为0,计算出现次数占总调查次数的频度作为家畜出现频度.计算公式为:
$$ H_{\rm{G}}={{t}}_i, $$ (1) $$ D_{\rm{L}}=\frac{{n}_{i}}{{S}_{i}} ,$$ (2) $$ {{F}}_{\rm{O}}=\frac{\displaystyle\sum _{i=1}{N}_{i}}{\displaystyle\sum _{i=1}{O}_{i}}, $$ (3) $$ I_{\rm{G}}=\frac{\left(H_{\rm{G}}+D_{\rm{L}}+F_{\rm{O}}\right)}{3}, $$ (4) 式中:ti表示每个样地所在牧场设立时间;ni表示家畜数量;Si表示样地所在牧场面积;Ni表示家畜出现次数;Oi表示调查次数.
1.8 数据处理与统计分析
利用非度量多维尺度法(non-metric multidimensional scaling, NMDS)和置换多元方差分析(PERMANOVA)对放牧区与非放牧区土壤理化性质和微生物组成整体构成的差异性进行评价.为了进一步评估土壤理化性质对干扰和环境因素的响应,我们建立了以土壤理化性质为响应变量,放牧干扰(放牧与否、放牧强度)和地形因子(海拔、坡度)为固定效应,取样点位置为随机效应的广义线性混合效应模型(generalized linear mixed models, GLMM),指数族选择高斯分布.GLMM模型的构建使用R语言中的glmmTMB程序包.
根据OTU数量计算相对丰度,将细菌和真菌中相对丰度<1%的归为其他,将相对丰度>5%的归为优势物种.采用Spearman相关性分析细菌和真菌相对丰度前5门的菌种与环境变量因子之间的关系.
使用基于OTU水平的NMDS进行β多样性研究,并通过置换999次的相似性分析(analysis of similarities, ANOSIM)对放牧区与非放牧区土壤微生物群落差异进行检验,并使用envfit函数通过999次置换检验微生物群落组成与放牧强度、地形变量和土壤理化性质等环境变量之间关系的显著性.以上所有计算过程在vegan包中完成.为了防止变量间存在多重共线性,我们计算了变量间皮尔逊相关系数(Pearson correlation coefficient)以及方差膨胀因子(variance inflation factor, VIF)(附件6、附件7),最终检验的变量包括海拔、坡度、放牧强度、电导率、pH及全氮、全钠、速效磷、可交换钾离子、可交换镁离子质量分数和植物丰富度,它们之间的相关性系数绝对值 < 0.7,VIF值 < 3.本研究数据分析利用R 4.2.1 (R Core Team, 2017)软件,作图使用ggplot2包.
2 结果
2.1 放牧对土壤理化性质的影响
NMDS和PERMANOVA结果显示,放牧区与非放牧区土壤理化性质构成显著不同(压力值为 0.21, P < 0.001) (附件2).不同土壤理化性质变量对放牧干扰的响应情况如表1所示,放牧区土壤电导率(Z = −1.970,P = 0.048)、密度(Z = −2.870,P = 0.005)及土壤全钙(Z = −2.900,P = 0.004)、土壤可交换K+(Z = −2.438,P = 0.015)与Ca2+(Z = −2.767,P = 0.006)质量分数均显著高于非放牧区,而土壤含水量(Z = 2.293,P = 0.022),土壤全钾(Z = 3.965,P < 0.001)、土壤可交换Na+(Z = 2.399,P < 0.016)质量分数均显著低于非放牧区,其他土壤特征指标(酸碱度及有机质、全氮、全磷、全钠、全镁、速效磷、速效钾、可交换Mg2+质量分数)并未受到放牧干扰的影响.
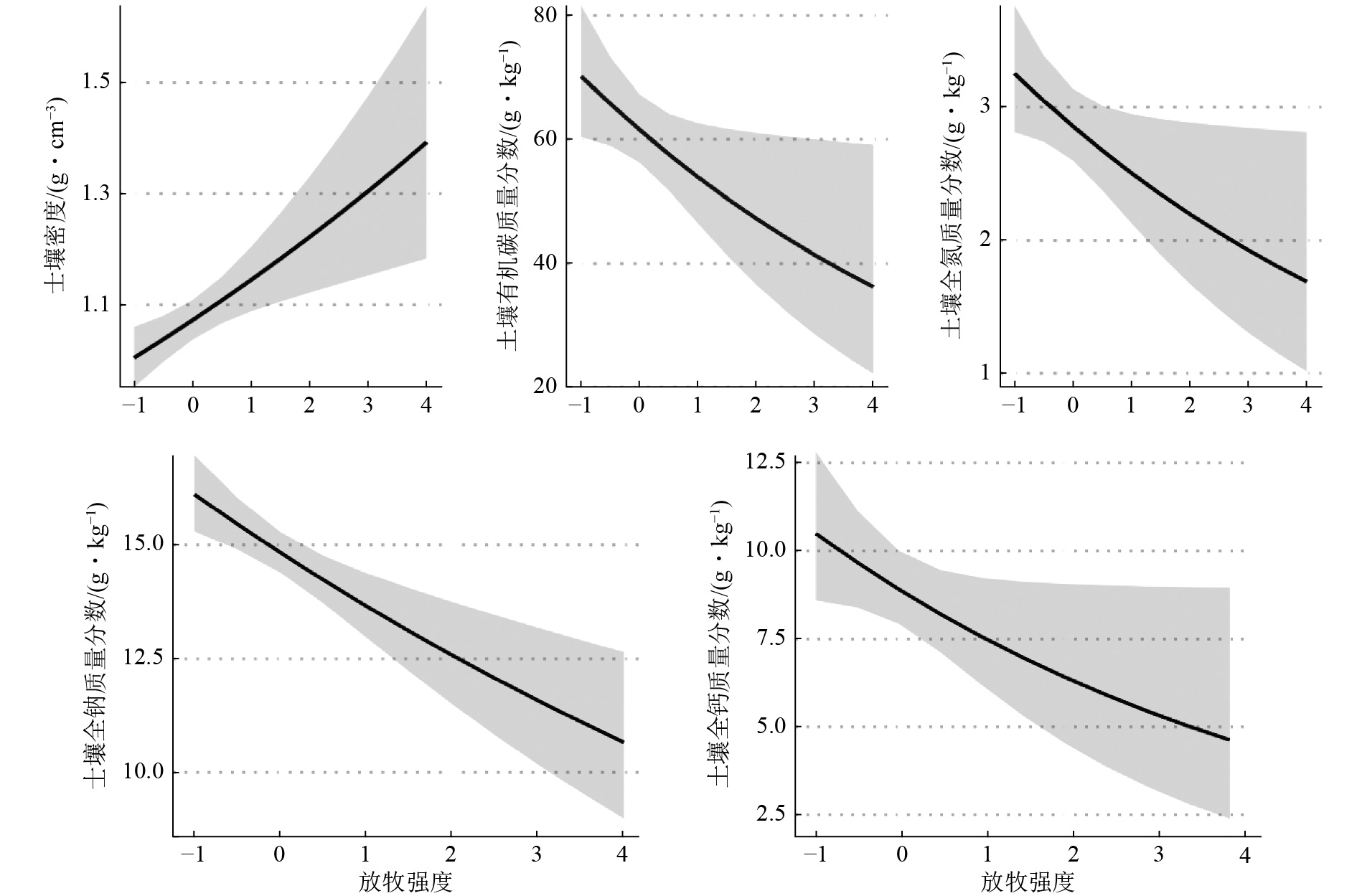
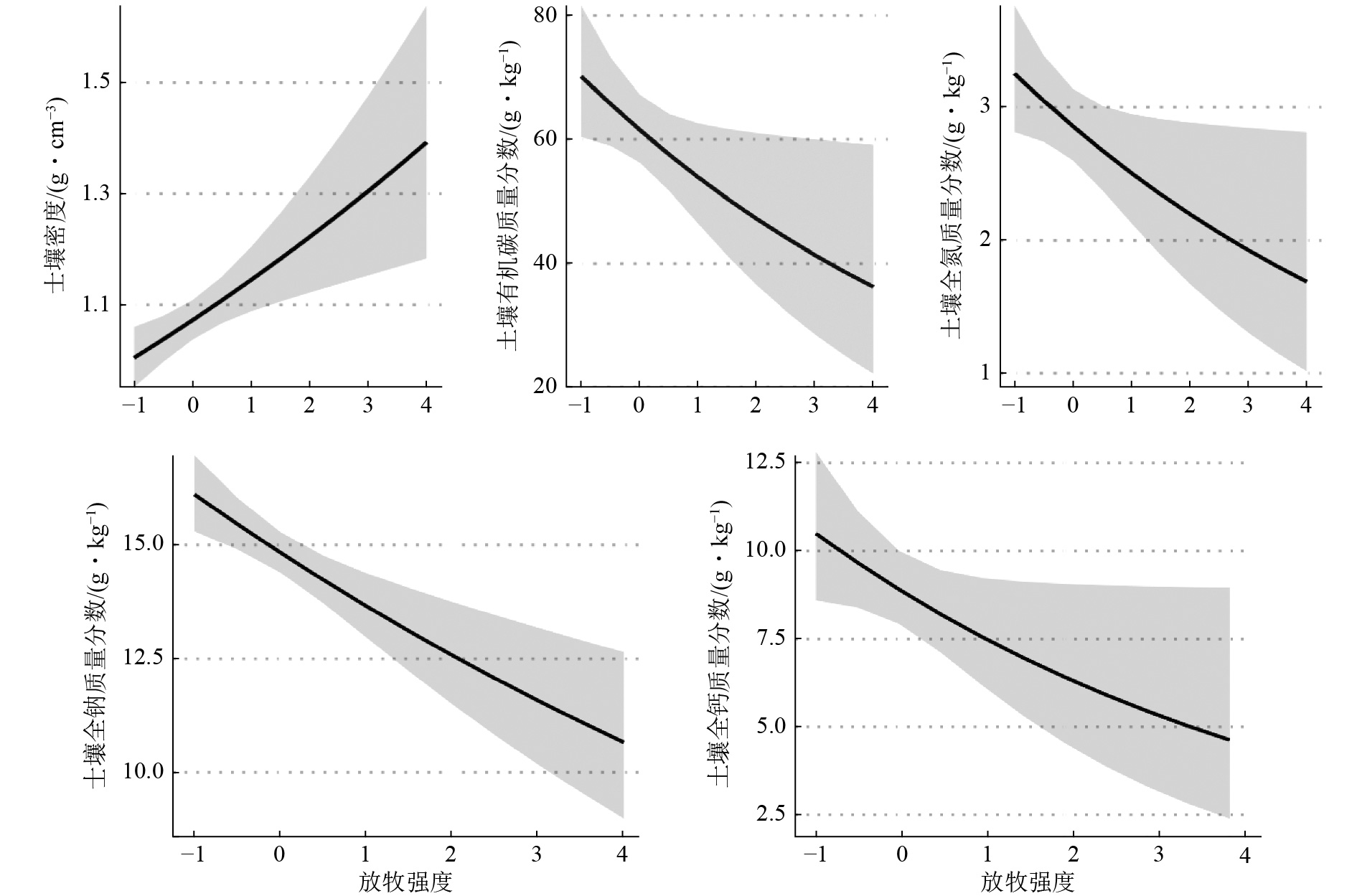
表 1 放牧区与非放牧区土壤理化性质(平均值±标准误)比较和广义混合效应模型显著性检验统计土壤因子 土壤理化性质 模型检验 放牧 非放牧 Z P 电导率/(S·m−1) 36.680 ± 1.59* 29.130 ± 1.72* −1.970 0.048 密度/(g·cm−1) 1.220 ± 0.03* 0.960 ± 0.02* −2.870 0.005 含水量/g·kg−1 31.840 ± 2.97* 51.390 ± 3.71* 2.293 0.022 酸碱度 5.520 ± 0.08 5.610 ± 0.08 0.290 0.774 有机碳质量分数/(g·kg−1) 69.400 ± 5.10 66.680 ± 3.76 −1.630 0.013 全氮质量分数/(g·kg−1) 3.050 ± 0.23 2.990 ± 0.14 −1.606 0.108 全磷质量分数/(g·kg−1) 0.770 ± 0.06 0.840 ± 0.04 1.046 0.295 全钾质量分数/(g·kg−1) 13.490 ± 0.46* 15.990 ± 0.38* 3.965 <0.001 全钠质量分数/(g·kg−1) 14.730 ± 0.43 15.380 ± 0.33 −1.280 0.200 全钙质量分数/(g·kg−1) 12.890 ± 1.33* 9.430 ± 0.85* −2.900 0.004 全镁质量分数/(g·kg−1) 9.970 ± 0.85 8.850 ± 0.78 −1.226 0.220 速效磷质量分数/(g·kg−1) 2.710 ± 0.49 3.500 ± 0.65 −0.283 0.777 速效钾质量分数/(g·kg−1) 135.050 ± 8.62 106.450 ± 7.43 −1.644 0.100 可交换K+质量分数/(g·kg−1) 0.520 ± 0.02* 0.390 ± 0.02* −2.438 0.015 可交换Na+质量分数/(g·kg−1) 0.800 ± 0.07* 1.250 ± 0.07* 2.399 0.016 可交换Ca2+质量分数/(g·kg−1) 11.630 ± 0.90* 9.290 ± 0.46* −2.767 0.006 可交换Mg2+质量分数/(g·kg−1) 2.360 ± 0.26 2.250 ± 0.10 0.254 0.799 注:*表示显著性差异(P < 0.05). 随放牧强度增加,土壤密度(Z = 3.600,P < 0.001)显著增加,而有机碳(Z = −2.180,P = 0.026)、全氮(Z = −2.150,P = 0.031)、全钠(Z = 3.900,P < 0.001)和全钙(Z = −2.001,P = 0.045)质量分数显著下降(图2 ),其他土壤特征指标(电导率、含水量、酸碱度和全磷、全钾、全镁、速效磷及可交换K+、Na+、Mg2+、Ca2+质量分数)并未受到放牧强度的影响(图3、附件8).
2.2 放牧对土壤微生物群落的影响
经过滤低质量序列和低丰度序列,获得2755547条细菌序列,按照样品最小序列数42262抽平获得2416741条细菌序列,划分为9488个OTUs,共41门139纲332目529科928属;获得1040555条真菌序列,按照样品最小序列数16672抽平后得到916986条真菌序列,划分为5676个OTUs,隶属于18门70纲165目375科856属.
选取ACE估计数、Chao1丰富度指数、Shannon多样性指数对放牧区和非放牧区的α多样性进行评估.放牧区的细菌群落Shannon多样性指数显著高于非放牧区;放牧区和非放牧区的真菌群落α多样性并无显著差异(附件4).
细菌和真菌群落组成见附件5.无论是放牧区还是非放牧区,从细菌门水平的相对丰度来看,主要是由变形菌门、放线菌门、酸杆菌门、绿弯菌门等优势门组成;从真菌门水平的相对丰度来看,担子菌门、子囊菌门、孢霉菌门等优势门是放牧区和非放牧区土壤真菌的主要组成部分.
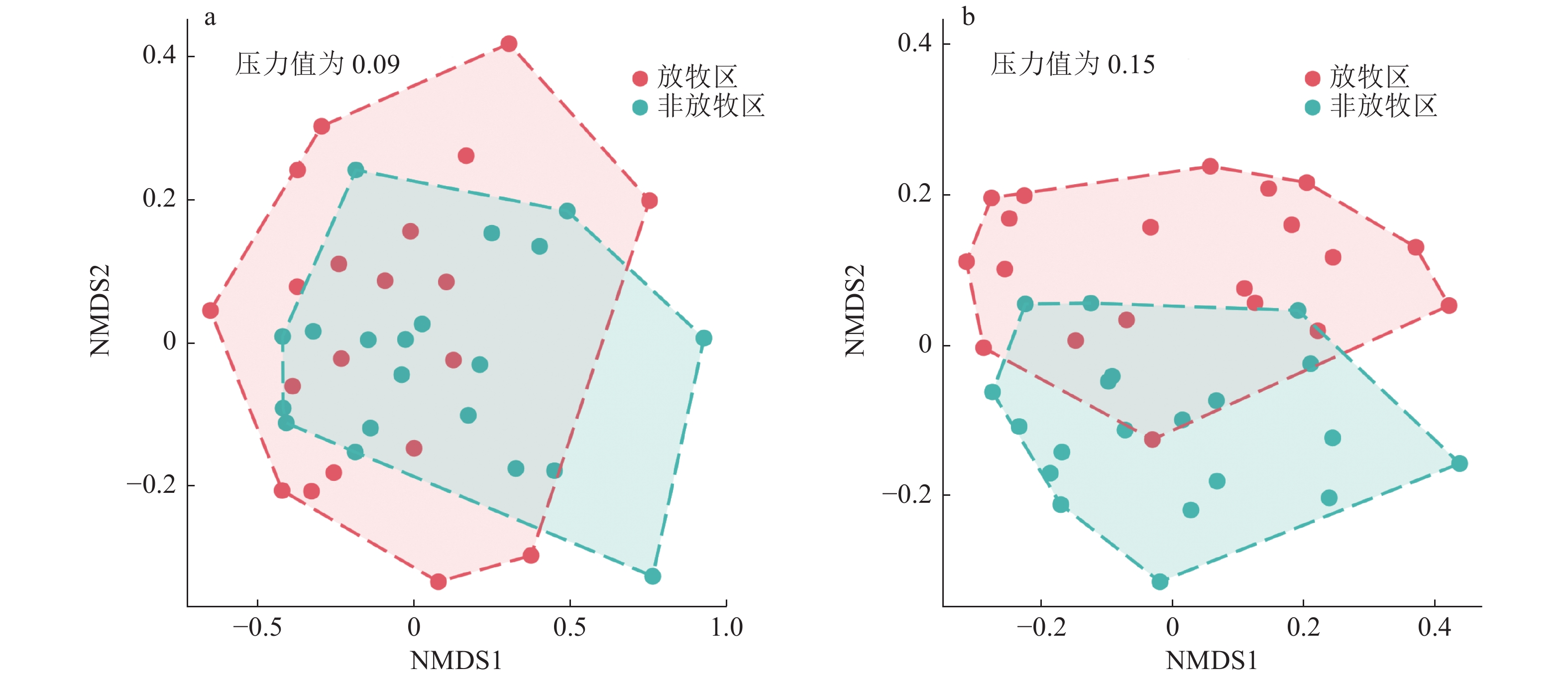
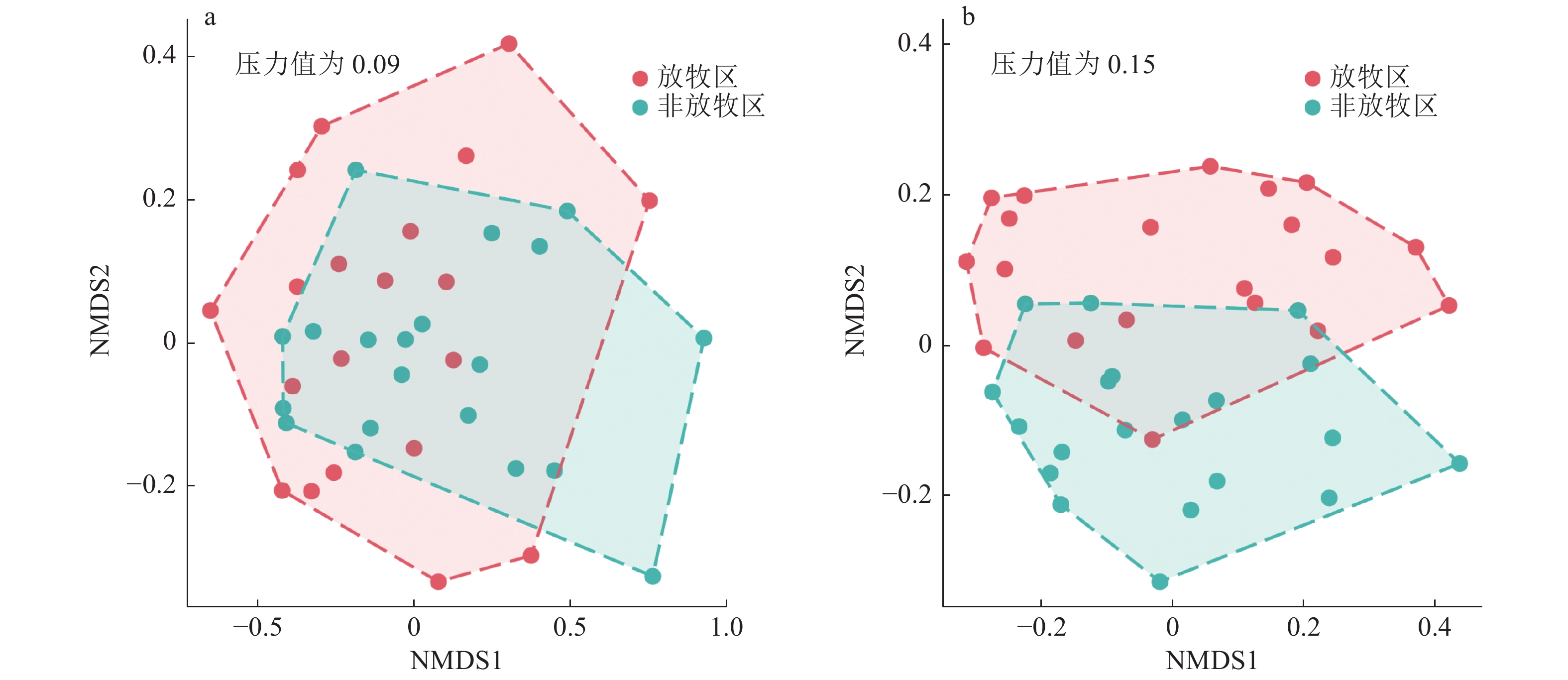
基于OTUs水平的Bray-Curtis距离的NMDS对放牧区与非放牧区的细菌和真菌群落分析(图4),并结合ANOSIM检验发现细菌(压力值为0.09, P = 0.021)(图4-a)和真菌(压力值为0.15, P = 0.042)(图4-b)之间组间差异大于组内, 表明放牧活动对细菌和真菌的群落组装分别具有显著影响.单一环境因子的显著性检验如表2所示,其中,坡度、电导率与植物丰富度显著影响细菌群落组成(P < 0.05),而放牧强度、海拔、坡度、电导率,以及全氮、速效磷质量分数和植物丰富度显著影响真菌群落组成.
表 2 细菌和真菌群落组成与环境变量的显著性检验变量 细菌 真菌 NMDS1 NMDS2 R2 P NMDS1 NMDS2 R2 P 放牧强度 −0.202 0.979 0.128 0.066 −0.085 0.996 0.384 < 0.001 海拔 0.136 −0.991 0.148 0.062 0.826 −0.564 0.217 0.008 坡度 −0.033 −0.999 0.338 0.001 1.000 −0.017 0.282 0.002 电导率 −0.463 0.886 0.219 0.010 −0.644 0.765 0.407 < 0.001 酸碱度 −0.849 0.528 0.091 0.179 −0.999 0.037 0.027 0.630 全氮质量分数 0.820 0.573 0.057 0.345 −0.327 −0.945 0.453 < 0.001 全钠质量分数 −0.487 −0.873 0.023 0.649 −0.788 −0.616 0.000 0.999 速效磷质量分数 −0.433 0.902 0.024 0.652 −0.848 −0.529 0.150 0.039 可交换K+质量分数 −0.986 −0.166 0.045 0.449 0.097 0.995 0.068 0.254 可交换Mg2+质量分数 −0.980 0.200 0.011 0.837 −0.980 −0.198 0.024 0.644 植物丰富度 −0.210 0.978 0.223 0.010 −0.823 0.567 0.270 0.009 进一步对优势物种门水平的相对丰度与环境变量进行Spearman相关性分析(表3)发现:放牧强度与变形菌门的相对丰度呈显著正相关;海拔与变形菌门、担子菌门的相对丰度呈显著正相关,与子囊菌门、孢霉菌门以及未被分类的真菌门的相对丰度呈显著负相关;坡度与担子菌门的相对丰度呈显著正相关,与子囊菌门、孢霉菌门、未被分类的真菌门及罗兹菌门的相对丰度呈显著负相关;电导率与变形菌门、担子菌门的相对丰度呈显著负相关,与放线菌门、绿弯菌门、子囊菌门、孢霉菌门、未被分类的真菌门及罗兹菌门的相对丰度呈显著正相关;全氮质量分数与未被分类的真菌门及罗兹菌门的相对丰度呈显著正相关;速效磷质量分数与担子菌门的相对丰度呈显著负相关,与子囊菌门、孢霉菌门、未被分类的真菌门及罗兹菌门的相对丰度呈显著正相关;植物丰富度与变形菌门与担子菌门的相对丰度呈显著负相关,与绿弯菌门、子囊菌门、孢霉菌门、未被分类的真菌门及罗兹菌门的相对丰度呈显著正相关.
表 3 细菌和真菌优势菌门的相对丰度与环境变量相关性分类 门 GRA ELE SLP SECP pH wT(N) wT(Na) wSA(P) wSE(K+) wSE(Mg2+) S 细菌 变形菌门 0.018* 0.032* 0.124 0.006* 0.644 0.401 0.126 0.947 0.263 0.879 −0.381* 酸杆菌门 0.324 0.281 0.191 0.129 0.492 0.709 0.956 0.212 0.383 0.849 −0.044 放线菌门 0.062 0.022 0.384 0.007* 0.604 0.082 0.159 0.792 0.979 0.442 0.073 绿弯菌门 0.160 0.092 0.057 0.015* 0.245 0.744 0.818 0.205 0.890 0.992 0.357* 疣微菌门 0.301 0.544 0.328 0.120 0.647 0.164 0.735 0.500 0.405 0.522 −0.009 真菌 担子菌门 −0.142 0.475* 0.575* −0.465* −0.108 −0.211 −0.154 −0.389* 0.030 −0.009 −0.370* 子囊菌门 0.198 −0.438* −0.534* 0.460* 0.122 0.111 0.163 0.345* −0.005 −0.078 0.353* 孢霉菌门 0.049 −0.388* −0.548* 0.446* 0.057 0.311 0.105 0.404* 0.043 0.116 0.339* 未被分类真菌 0.023 −0.493* −0.569* 0.468* 0.057 0.365* 0.158 0.487* 0.013 0.120 0.393* 罗兹菌门 −0.053 −0.301* −0.437* 0.235 0.101 0.381* 0.027 0.433* −0.143 −0.128 0.313* 注:GRA,放牧强度;ELE,海拔;SLP, 坡度; SECP,土壤电导率;pH,酸碱度;wT(N),土壤全氮质量分数;wT(Na),土壤全钠质量分数;wSA(P),土壤速效磷质量分数;wSE(K+),土壤可交换K+质量分数;wSE(Mg2+),土壤可交换Mg2+质量分数;S, 植物丰富度.*表示显著性水平P < 0.05. 3 讨论
3.1 放牧对土壤理化性质的影响
土壤理化性质对水、空气、养分等土壤资源动态以及植物生长、土壤生物组成和群落形成具有重要影响,并最终影响森林生态系统的服务功能[47].因此,了解土壤理化性质对放牧干扰的响应具有重要意义.东北虎豹公园永久监测样地上量化了土壤理化性质对放牧强度的响应.研究结果表明,放牧对东北虎豹国家公园土壤理化性质造成了不同程度的影响.对土壤理化指标而言,放牧显著影响了土壤电导率、密度及含水量,放牧区内的电导率与密度均显著高于非放牧区,而土壤含水量则显著低于非放牧区,这与Frolla等[48]的研究结果相符.
土壤密度是土壤紧实度的指标之一,它与土壤孔隙度和渗透率密切相关.土壤密度可以反映土壤结构的好坏,影响着土壤中水、肥、气、热等肥力因素的变化和协调.本研究结果中放牧对于土壤密度的影响是最明显的,说明放牧使土壤理化性状改变,土壤密度降低代表着牲畜的践踏作用和放牧伴随的人类活动使得土壤失去了大中等孔隙,这可能是导致国家公园放牧区内土壤密度显著高于非放牧区的原因,这与多数研究结果一致[48−50],而且由于累积效应的影响,不同的放牧强度对于密度的影响是不同的[11],合理的放牧可以提高渗透率和保持土壤肥力,但过度放牧会导致土壤空隙的消失,使得土壤密度升高,严重限制了土壤的渗透能力以及保水保肥能力[51−52];放牧区内的土壤含水量显著地低于非放牧区,表明国家公园的放牧行为已经严重过载,导致了土地退化,生产力下降,并且放牧会增大地表的裸露程度,地表蒸发随之增大,土壤水分不易保持,进一步降低土壤含水量[49];此外,土壤电导率反映了土壤中可溶性离子浓度,放牧区电导率升高表明土壤有盐碱化的趋势[50],进一步证实了公园内的过度放牧及人类活动降低了土壤品质,不利于植被恢复,甚至有可能会引发盐害.
3.2 放牧对土壤养分池的影响
土壤中包含植物生长必需的矿质营养元素,这些元素是森林生产力的首要限制因素,也是决定群落结构的重要因子.放牧对土壤矿质元素的影响主要来自2方面: 1)牲畜活动伴随的尿液与粪便堆积而产生的养分累加效应[24,27,53],牲畜摄取的食物中仅有小部分(3%~30%)营养元素被动物本身利用,其余大部分则以粪便和尿液的形式排出,因而牲畜粪便通常富含营养元素,这些养分被土壤吸收并且随着面积粪便数量增加而显著升高,为植被生长提供长期的营养供应[54]; 2)牲畜的践踏作用和对地上植被的啃食破坏引起的反馈作用,导致土壤养分流失[52].
关于放牧对土壤养分池含量的影响一直没有定论:一种观点认为放牧可以增加土壤养分元素含量;第2种观点则反之;第3观点认为放牧与土壤养分含量之间的关系复杂,不能从单一因素角度考虑[52,55−56].我们的研究结果显示,国家公园土壤全钙、可交换K+和Ca2+质量分数因受到放牧干扰都显著升高,这有可能是由于家畜的践踏作用减小了凋落物颗粒大小,促进凋落物的分解,有利于养分融入土壤,加速了土壤钾与钙的积累.同时,家畜排泄物的堆积与降解也是矿质元素反哺土壤的重要机制,这一机制加速了群落养分循环,补充了土壤中矿质元素的存储[57−59].放牧使得森林植被也遭到破坏,这种变化会随着营养级联传递到土壤养分池[3,11],土壤矿质元素的积累是收支差异的结果,植物生物量的显著下降,导致植物吸收能力下降,造成土壤矿质元素的冗余[60].放牧区内的全钾质量分数代表了土壤中钾元素的总储量,对比结果证实由于土壤结构被破坏,虽然动物粪便分解的反馈效应补充了一些矿质元素,但整体而言,放牧干扰还是造成了土壤钾元素的流失.由于放牧地区家畜活动强烈,加速了土壤可溶性矿质元素离子的循环分解,这可能是导致土壤可交换钠质量分数在放牧区表现较低水平的原因[52].
3.3 放牧对微生物群落组成的影响
土壤微生物是土壤生态系统的重要组成部分,它们作为分解者,参与土壤养分分解、循环和转化的调控过程,在植物-土壤反馈过程中发挥着重要作用[18,28,30,55].研究结果表明:放牧干扰显著改变了土壤微生物的群落组成(图4);同时地形和土壤养分也是形成和维持土壤微生物群落构成的主要影响因素[51,60−63].
东北虎豹国家公园内长期的林下放牧,已经对土壤微生物群落产生了显著的影响.我们的研究结果显示,细菌和真菌的β多样性在放牧区与非放牧区之间差异显著(图4).放牧地区的土壤通常有机质丰富、植物根系众多、通气状况良好且水分适中,有利于土壤微生物的生存和繁殖,并且牲畜的践踏、排泄等行为也会促进土壤微生物的活动[64].但随着放牧强度的进一步增强,地上植被、土壤理化性质和营养物质在时空上的异质性发生显著改变,促使土壤微生物生存环境发生变化,往往导致微生物群落结构发生变化[65].不同分类水平的微生物类群对放牧干扰的响应表现出很大的差异.在门水平上,只有变形菌门受到放牧强度的影响.变形菌门是世界上最普遍的菌门[66],它们多为抗逆性较强的寡营养类群且可以分泌胞外多糖,这可以黏结沙粒,避免土壤发生水土流失,保持土壤养分[67];在氮元素受限制的土壤中,它们还可以起到固氮的作用[68],为植物提供生长所需的养分.
土壤为微生物群落提供了生存环境,因此,土地利用方式改变引起的土壤理化性质变化会导致微生物组成也随之变化[62,69].研究结果表明,无论是土壤细菌还是真菌群落,土壤理化性质都是维持其群落组成的主要影响因素.电导率、全氮、速效磷显著影响真菌群落组成,土壤电导率是改变细菌群落组成的显著变量,说明土壤理化性质也是决定国家公园土壤微生物群落组成的重要因素.微生物群落受土壤微生境的影响,与土壤自身属性有着强烈的相关性 [22];而土壤微生物也是土壤生态系统的组分之一,几乎所有的土壤变化过程都直接或间接地与土壤微生物有关,因此土壤养分状况是与细菌和真菌群落组成变化密切相关的因素[27].
除了土壤理化性质,地形因素和植被也是调控土壤微生物群落组成的主要因素.海拔的变化造成了生态环境的差异,导致土壤微生物的环境过滤和生态位分离[70].近年来,关于海拔对土壤微生物的研究有许多,但并未得出一致的结论.例如:Whitaker等[60]研究发现安第斯山脉土壤中细菌和真菌的丰富度随着海拔的增加而升高;Wagai等[61]研究发现,随着海拔的升高,细菌和真菌多样性下降;Djukic等[62]发现,细菌的多样性并未随着海拔增加而表现出显著的变化规律;Siles等[63]发现,随着海拔的增加细菌相对丰度也会随之增加.因此,海拔对土壤微生物的影响是多元的:1)海拔会对植被造成一定影响,而植被会通过反馈调节进而影响土壤微生物;2)土壤理化性质对海拔变化十分敏感,间接地改变微生物的群落结构[71];3)海拔升高导致温度降低,也会对土壤微生物的活动产生一定的影响[70].不同海拔、坡度条件下所形成的森林小气候存在明显差异,不同气候下的降水及空气温、湿度等差异较大.物种的扩散与定植需要土壤提供合适的温度、湿度与养分,因此,地形因素是国家公园土壤微生物群落物种分布差异的主要原因之一,这与大多数放牧背景下土壤微生物和动物群落分布研究结果相一致[28,48,72].此外,放牧引起的植物群落结构和物种组成的改变:一方面会通过营养级联传递到土壤,进而影响土壤生物群落组成[12,29];另一方面,真菌中还有大量的菌根真菌, 与植物形成共生关系, 协助植物获取养分特别是氮和磷[73−74].所以国家公园植物的丰富度变化会显著影响土壤微生物的群落结构.
4 结论
本文研究了东北虎豹国家公园长期林下放牧对土壤理化性质和微生物群落结构的影响.结果表明,家畜的活动显著增加了土壤密度和电导率,降低了土壤含水量,表明家畜的活动和践踏作用可能已经破坏了土壤结构,降低了土壤保水能力.放牧干扰使得土壤中全钙、可交换K+和Ca2+质量分数显著升高,这说明家畜的活动以及粪便的积累与降解可能加速了土壤矿质元素的循环,增加了土壤养分池的含量.放牧干扰本身以及通过对土壤理化性质的影响,进一步影响了土壤微生物的群落结构.林下放牧对生态系统结构、功能的影响是复杂的,涉及多个营养级与生态过程,未来需要加强放牧对土壤食物网中各生物类群以及相关的地下生态过程和功能方面的研究,为东北虎豹国家公园生态系统功能的恢复和动物栖息地质量的提升提供科学依据.
数据分享:附件1~8见期刊网站.数据链接:http://www.bnujournal.com/article/doi/10.12202/j.0476-0301.2023140.
-
表 1 放牧区与非放牧区土壤理化性质(平均值±标准误)比较和广义混合效应模型显著性检验统计
土壤因子 土壤理化性质 模型检验 放牧 非放牧 Z P 电导率/(S·m−1) 36.680 ± 1.59* 29.130 ± 1.72* −1.970 0.048 密度/(g·cm−1) 1.220 ± 0.03* 0.960 ± 0.02* −2.870 0.005 含水量/g·kg−1 31.840 ± 2.97* 51.390 ± 3.71* 2.293 0.022 酸碱度 5.520 ± 0.08 5.610 ± 0.08 0.290 0.774 有机碳质量分数/(g·kg−1) 69.400 ± 5.10 66.680 ± 3.76 −1.630 0.013 全氮质量分数/(g·kg−1) 3.050 ± 0.23 2.990 ± 0.14 −1.606 0.108 全磷质量分数/(g·kg−1) 0.770 ± 0.06 0.840 ± 0.04 1.046 0.295 全钾质量分数/(g·kg−1) 13.490 ± 0.46* 15.990 ± 0.38* 3.965 <0.001 全钠质量分数/(g·kg−1) 14.730 ± 0.43 15.380 ± 0.33 −1.280 0.200 全钙质量分数/(g·kg−1) 12.890 ± 1.33* 9.430 ± 0.85* −2.900 0.004 全镁质量分数/(g·kg−1) 9.970 ± 0.85 8.850 ± 0.78 −1.226 0.220 速效磷质量分数/(g·kg−1) 2.710 ± 0.49 3.500 ± 0.65 −0.283 0.777 速效钾质量分数/(g·kg−1) 135.050 ± 8.62 106.450 ± 7.43 −1.644 0.100 可交换K+质量分数/(g·kg−1) 0.520 ± 0.02* 0.390 ± 0.02* −2.438 0.015 可交换Na+质量分数/(g·kg−1) 0.800 ± 0.07* 1.250 ± 0.07* 2.399 0.016 可交换Ca2+质量分数/(g·kg−1) 11.630 ± 0.90* 9.290 ± 0.46* −2.767 0.006 可交换Mg2+质量分数/(g·kg−1) 2.360 ± 0.26 2.250 ± 0.10 0.254 0.799 注:*表示显著性差异(P < 0.05). 表 2 细菌和真菌群落组成与环境变量的显著性检验
变量 细菌 真菌 NMDS1 NMDS2 R2 P NMDS1 NMDS2 R2 P 放牧强度 −0.202 0.979 0.128 0.066 −0.085 0.996 0.384 < 0.001 海拔 0.136 −0.991 0.148 0.062 0.826 −0.564 0.217 0.008 坡度 −0.033 −0.999 0.338 0.001 1.000 −0.017 0.282 0.002 电导率 −0.463 0.886 0.219 0.010 −0.644 0.765 0.407 < 0.001 酸碱度 −0.849 0.528 0.091 0.179 −0.999 0.037 0.027 0.630 全氮质量分数 0.820 0.573 0.057 0.345 −0.327 −0.945 0.453 < 0.001 全钠质量分数 −0.487 −0.873 0.023 0.649 −0.788 −0.616 0.000 0.999 速效磷质量分数 −0.433 0.902 0.024 0.652 −0.848 −0.529 0.150 0.039 可交换K+质量分数 −0.986 −0.166 0.045 0.449 0.097 0.995 0.068 0.254 可交换Mg2+质量分数 −0.980 0.200 0.011 0.837 −0.980 −0.198 0.024 0.644 植物丰富度 −0.210 0.978 0.223 0.010 −0.823 0.567 0.270 0.009 表 3 细菌和真菌优势菌门的相对丰度与环境变量相关性
分类 门 GRA ELE SLP SECP pH wT(N) wT(Na) wSA(P) wSE(K+) wSE(Mg2+) S 细菌 变形菌门 0.018* 0.032* 0.124 0.006* 0.644 0.401 0.126 0.947 0.263 0.879 −0.381* 酸杆菌门 0.324 0.281 0.191 0.129 0.492 0.709 0.956 0.212 0.383 0.849 −0.044 放线菌门 0.062 0.022 0.384 0.007* 0.604 0.082 0.159 0.792 0.979 0.442 0.073 绿弯菌门 0.160 0.092 0.057 0.015* 0.245 0.744 0.818 0.205 0.890 0.992 0.357* 疣微菌门 0.301 0.544 0.328 0.120 0.647 0.164 0.735 0.500 0.405 0.522 −0.009 真菌 担子菌门 −0.142 0.475* 0.575* −0.465* −0.108 −0.211 −0.154 −0.389* 0.030 −0.009 −0.370* 子囊菌门 0.198 −0.438* −0.534* 0.460* 0.122 0.111 0.163 0.345* −0.005 −0.078 0.353* 孢霉菌门 0.049 −0.388* −0.548* 0.446* 0.057 0.311 0.105 0.404* 0.043 0.116 0.339* 未被分类真菌 0.023 −0.493* −0.569* 0.468* 0.057 0.365* 0.158 0.487* 0.013 0.120 0.393* 罗兹菌门 −0.053 −0.301* −0.437* 0.235 0.101 0.381* 0.027 0.433* −0.143 −0.128 0.313* 注:GRA,放牧强度;ELE,海拔;SLP, 坡度; SECP,土壤电导率;pH,酸碱度;wT(N),土壤全氮质量分数;wT(Na),土壤全钠质量分数;wSA(P),土壤速效磷质量分数;wSE(K+),土壤可交换K+质量分数;wSE(Mg2+),土壤可交换Mg2+质量分数;S, 植物丰富度.*表示显著性水平P < 0.05. -
[1] BARNOSKY A D,HADLY E A,BASCOMPTE J,et al. Approaching a state shift in Earth’s biosphere[J]. Nature,2012,486(7401):52 doi: 10.1038/nature11018
[2] BRODIE J F,WILLIAMS S,GARNER B. The decline of mammal functional and evolutionary diversity worldwide[J]. Proceedings of the National Academy of Sciences of the United States of America,2021,118(3):e1921849118
[3] FILAZZOLA A,BROWN C,DETTLAFF M A,et al. The effects of livestock grazing on biodiversity are multi-trophic:a meta-analysis[J]. Ecology Letters,2020,23(8):1298 doi: 10.1111/ele.13527
[4] SCHIELTZ J M,RUBENSTEIN D I. Evidence based review:positive versus negative effects of livestock grazing on wildlife. What do we really know?[J]. Environmental Research Letters,2016,11(11):113003 doi: 10.1088/1748-9326/11/11/113003
[5] KIMBALL S,SCHIFFMAN P M. Differing effects of cattle grazing on native and alien plants[J]. Conservation Biology,2003,17(6):1681 doi: 10.1111/j.1523-1739.2003.00205.x
[6] TOTH E,DEAK B,VALKO O,et al. Livestock type is more crucial than grazing intensity:traditional cattle and sheep grazing in short-grass steppes[J]. Land Degradation and Development,2018,29(2):231 doi: 10.1002/ldr.2514
[7] KOHL M T,KRAUSMAN P R,KUNKEL K,et al. Bison versus cattle:are they ecologically synonymous?[J]. Rangeland Ecology & Management,2013,66(6):721
[8] YUAN Z Y,JIAO F,LI Y H,et al. Anthropogenic disturbances are key to maintaining the biodiversity of grasslands[J]. Scientific Reports,2016,6:22132 doi: 10.1038/srep22132
[9] ROBINSON T P,WILLIAM WINT G R,CONCHEDDA G,et al. Mapping the global distribution of livestock[J]. PLoS One,2014,9(5):e96084 doi: 10.1371/journal.pone.0096084
[10] LI B V,JIANG B K. Responses of forest structure,functions,and biodiversity to livestock disturbances:a global meta-analysis[J]. Global Change Biology,2021,27(19):4745 doi: 10.1111/gcb.15781
[11] LAI L M,KUMAR S. A global meta-analysis of livestock grazing impacts on soil properties[J]. PLoS One,2020,15(8):e0236638 doi: 10.1371/journal.pone.0236638
[12] BYRNES R C,EASTBURN D J,TATE K W,et al. A global meta-analysis of grazing impacts on soil health indicators[J]. Journal of Environmental Quality,2018,47(4):758 doi: 10.2134/jeq2017.08.0313
[13] KARLEN D L,MAUSBACH M J,DORAN J W,et al. Soil quality:a concept,definition,and framework for evaluation (a guest editorial)[J]. Soil Science Society of America Journal,1997,61(1):4 doi: 10.2136/sssaj1997.03615995006100010001x
[14] TANG L,ZHONG L,XUE K,et al. Warming counteracts grazing effects on the functional structure of the soil microbial community in a Tibetan grassland[J]. Soil Biology and Biochemistry,2019,134:113 doi: 10.1016/j.soilbio.2019.02.018
[15] FENETAHUN Y,YOU Y A,XU X W,et al. Impact of grazing intensity on soil properties in teltele rangeland,Ethiopia[J]. Frontiers in Environmental Science,2021,9:664104 doi: 10.3389/fenvs.2021.664104
[16] ZHANG B,THOMAS B W,BECK R,et al. Labile soil organic matter in response to long-term cattle grazing on sloped rough fescue grassland in the foothills of the Rocky Mountains,Alberta[J]. Geoderma,2018,318:9 doi: 10.1016/j.geoderma.2017.12.019
[17] ZHANG P,YANG Z C,WU J H. Livestock grazing promotes ecosystem multifunctionality of a coastal salt marsh [J]. Journal of Applied Ecology,2021,58(10):2124
[18] XU W L,ZHAO M M,REN R R,et al. Grazing amplifies the stimulating effects of N addition on N2O emissions in a temperate meadow steppe[J]. Agriculture,Ecosystems & Environment,2022,339:108143
[19] ELDRIDGE D J,DELGADO-BAQUERIZO M,TRAVERS S K,et al. Do grazing intensity and herbivore type affect soil health?: insights from a semi-arid productivity gradient[J]. Journal of Applied Ecology,2017,54(3):976 doi: 10.1111/1365-2664.12834
[20] IRSHAD U,VILLENAVE C,BRAUMAN A,et al. Grazing by nematodes on rhizosphere bacteria enhances nitrate and phosphorus availability to Pinus pinaster seedlings[J]. Soil Biology and Biochemistry,2011,43(10):2121 doi: 10.1016/j.soilbio.2011.06.015
[21] GEISEN S,WALL D H,VAN DER PUTTEN W H. Challenges and opportunities for soil biodiversity in the anthropocene[J]. Current Biology,2019,29(19):R1036 doi: 10.1016/j.cub.2019.08.007
[22] DELGADO-BAQUERIZO M,OLIVERIO A M,BREWER T E,et al. A global atlas of the dominant bacteria found in soil[J]. Science,2018,359(6373):320 doi: 10.1126/science.aap9516
[23] ROUSK J,BÅÅTH E. Growth of saprotrophic fungi and bacteria in soil[J]. FEMS Microbiology Ecology,2011,78(1):17 doi: 10.1111/j.1574-6941.2011.01106.x
[24] TANG S M,WANG K,XIANG Y Z,et al. Heavy grazing reduces grassland soil greenhouse gas fluxes:a global meta-analysis[J]. Science of the Total Environment,2019,654:1218 doi: 10.1016/j.scitotenv.2018.11.082
[25] LIU N,KAN H M,YANG G W,et al. Changes in plant,soil,and microbes in a typical steppe from simulated grazing:explaining potential change in soil C[J]. Ecological Monographs,2015,85(2):269 doi: 10.1890/14-1368.1
[26] KAUFFMAN J B,THORPE A S,JACK BROOKSHIRE E N. Livestock exclusion and belowground ecosystem responses in riparian meadows of eastern Oregon[J]. Ecological Applications,2004,14(6):1671 doi: 10.1890/03-5083
[27] ELDRIDGE D J,DELGADO-BAQUERIZO M,TRAVERS S K,et al. Competition drives the response of soil microbial diversity to increased grazing by vertebrate herbivores[J]. Ecology,2017,98(7):1922 doi: 10.1002/ecy.1879
[28] ZHAO F Z,REN C J,SHELTON S,et al. Grazing intensity influence soil microbial communities and their implications for soil respiration[J]. Agriculture,Ecosystems & Environment,2017,249:50
[29] MUELLER P,GRANSE D,NOLTE S,et al. Top-down control of carbon sequestration:grazing affects microbial structure and function in salt marsh soils[J]. Ecological Applications,2017,27(5):1435 doi: 10.1002/eap.1534
[30] HUHE,CHEN X J,HOU F J,et al. Bacterial and fungal community structures in loess plateau grasslands with different grazing intensities[J]. Frontiers in Microbiology,2017,8:606 doi: 10.3389/fmicb.2017.00606
[31] STRICKLAND M S,ROUSK J. Considering fungal:bacterial dominance in soils - methods,controls,and ecosystem implications[J]. Soil Biology and Biochemistry,2010,42(9):1385 doi: 10.1016/j.soilbio.2010.05.007
[32] WANG T M,ANDREW ROYLE J,SMITH J L D,et al. Living on the edge:opportunities for Amur tiger recovery in China[J]. Biological Conservation,2018,217:269 doi: 10.1016/j.biocon.2017.11.008
[33] FENG J W,SUN Y F,LI H L,et al. Assessing mammal species richness and occupancy in a Northeast Asian temperate forest shared by cattle[J]. Diversity and Distributions,2021,27(5):857 doi: 10.1111/ddi.13237
[34] FENG R N,LÜ X Y,XIAO W H,et al. Effects of free-ranging livestock on sympatric herbivores at fine spatiotemporal scales[J]. Landscape Ecology,2021,36(5):1441 doi: 10.1007/s10980-021-01226-6
[35] WANG T M,FENG L M,YANG H T,et al. A science-based approach to guide Amur leopard recovery in China[J]. Biological Conservation,2017,210:47 doi: 10.1016/j.biocon.2016.03.014
[36] 王乐,冯佳伟,TSEVEEN A,等. 森林放牧对东北虎豹国家公园东部有蹄类动物灌草层食物资源的影响[J]. 兽类学报,2019,39(4):386 doi: 10.16829/j.slxb.150303 [37] 王乐,杨丽萌,赛九玛,等. 东北虎豹国家公园东部有蹄类栖息地的灌草层食物资源的量与质[J]. 兽类学报,2019,39(4):373 doi: 10.16829/j.slxb.150302 [38] 王天明,冯利民,杨海涛,等. 东北虎豹生物多样性红外相机监测平台概述[J]. 生物多样性,2020,28(9):1059 [39] ZHU A X,YANG L,LI B L,et al. Construction of membership functions for predictive soil mapping under fuzzy logic[J]. Geoderma,2010,155(3/4):164
[40] GAO B B,PAN Y C,CHEN Z Y,et al. A spatial conditioned Latin hypercube sampling method for mapping using ancillary data[J]. Transactions in GIS,2016,20(5):735 doi: 10.1111/tgis.12176
[41] 鲍士旦. 土壤农化分析[M]. 3版. 北京:中国农业出版社,2000 [42] MAGOČ T,SALZBERG S L. FLASH:fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics,2011,27(21):2957 doi: 10.1093/bioinformatics/btr507
[43] EDGAR R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods,2013,10(10):996 doi: 10.1038/nmeth.2604
[44] STACKEBRANDT E,GOEBEL B M. Taxonomic note:a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J]. International Journal of Systematic and Evolutionary Microbiology,1994,44(4):846 doi: 10.1099/00207713-44-4-846
[45] ALLEN V G,BATELLO C,BERRETTA E J,et al. An international terminology for grazing lands and grazing animals[J]. Grass and Forage Science,2011,66(1):2 doi: 10.1111/j.1365-2494.2010.00780.x
[46] WAUDBY H P,PETIT S,ROBINSON G. Pastoralists’ knowledge of plant palatability and grazing indicators in an arid region of South Australia[J]. The Rangeland Journal,2013,35(4):445 doi: 10.1071/RJ13021
[47] TRIGO C B,VILLAGRA P E,COWPER COLES P,et al. Can livestock exclusion affect understory plant community structure?: an experimental study in the dry Chaco forest,Argentina[J]. Forest Ecology and Management,2020,463:118014 doi: 10.1016/j.foreco.2020.118014
[48] FROLLA F,APARICIO V,COSTA J L,et al. Soil physical properties under different cattle stocking rates on Mollisols in the Buenos Aires Province,Argentina[J]. Geoderma Regional,2018,14:e00177 doi: 10.1016/j.geodrs.2018.e00177
[49] MOFIDI M,RASHTBARI M,ABBASPOUR H,et al. Impact of grazing on chemical,physical and biological properties of soils in the mountain rangelands of Sahand,Iran[J]. The Rangeland Journal,2012,34(3):297 doi: 10.1071/RJ11087
[50] HIERNAUX P,BIELDERS C L,VALENTIN C,et al. Effects of livestock grazing on physical and chemical properties of sandy soils in Sahelian rangelands[J]. Journal of Arid Environments,1999,41(3):231 doi: 10.1006/jare.1998.0475
[51] GARCIA M R L,SAMPAIO A A M,NAHAS E. Impact of different grazing systems for bovine cattle on the soil microbiological and chemical characteristics[J]. Revista Brasileira De Zootecnia,2011,40(7):1568 doi: 10.1590/S1516-35982011000700024
[52] ZHANG B,BECK R,PAN Q M,et al. Soil physical and chemical properties in response to long-term cattle grazing on sloped rough fescue grassland in the foothills of the Rocky Mountains,Alberta[J]. Geoderma,2019,346:75 doi: 10.1016/j.geoderma.2019.03.029
[53] YAN Y L,GANJURJAV H,HU G Z,et al. Nitrogen deposition induced significant increase of N2O emissions in an dry alpine meadow on the central Qinghai-Tibetan Plateau[J]. Agriculture,Ecosystems & Environment,2018,265:45
[54] SMET M,WARD D. Soil quality gradients around water-points under different management systems in a semi-arid savanna,South Africa[J]. Journal of Arid Environments,2006,64(2):251 doi: 10.1016/j.jaridenv.2005.04.014
[55] BAUER A,COLE C V,BLACK A L. Soil property comparisons in virgin grasslands between grazed and nongrazed management systems[J]. Soil Science Society of America Journal,1987,51(1):176 doi: 10.2136/sssaj1987.03615995005100010037x
[56] FRANK D A. Ungulate and topographic control of nitrogen:phosphorus stoichiometry in a temperate grassland; soils,plants and mineralization rates[J]. Oikos,2008,117(4):591 doi: 10.1111/j.0030-1299.2008.16220.x
[57] JAVADI S A,JAFARI M,AZARNIVAND H,et al. Investigation on grazing effects upon soil parameters at lar summer rangeland[J]. Journal of Agricultural Sciences,2006,11(4):71
[58] ZAREKIA S,JAFARI M,ARZANI H,et al. Grazing effects on some of the physical and chemical properties of soil[J]. World Applied Sciences Journal,2012,20(2):205
[59] RODRIGO-COMINO J,SENCIALES J M,CERDÀ A,et al. The multidisciplinary origin of soil geography:a review[J]. Earth-Science Reviews,2018,177:114 doi: 10.1016/j.earscirev.2017.11.008
[60] WHITAKER J,OSTLE N,NOTTINGHAM A T,et al. Microbial community composition explains soil respiration responses to changing carbon inputs along an Andes-to-Amazon elevation gradient[J]. The Journal of Ecology,2014,102(4):1058 doi: 10.1111/1365-2745.12247
[61] WAGAI R,KITAYAMA K,SATOMURA T,et al. Interactive influences of climate and parent material on soil microbial community structure in Bornean tropical forest ecosystems[J]. Ecological Research,2011,26(3):627 doi: 10.1007/s11284-011-0822-7
[62] DJUKIC I,ZEHETNER F,MENTLER A,et al. Microbial community composition and activity in different Alpine vegetation zones[J]. Soil Biology and Biochemistry,2010,42(2):155 doi: 10.1016/j.soilbio.2009.10.006
[63] SILES J A,MARGESIN R. Abundance and diversity of bacterial,archaeal,and fungal communities along an altitudinal gradient in alpine forest soils:what are the driving factors?[J]. Microbial Ecology,2016,72(1):207 doi: 10.1007/s00248-016-0748-2
[64] CINGOLANI A M,POSSE G,COLLANTES M B. Plant functional traits,herbivore selectivity and response to sheep grazing in Patagonian steppe grasslands[J]. Journal of Applied Ecology,2005,42(1):50 doi: 10.1111/j.1365-2664.2004.00978.x
[65] YANG F,NIU K C,COLLINS C G,et al. Grazing practices affect the soil microbial community composition in a Tibetan alpine meadow[J]. Land Degradation & Development,2019,30(1):49
[66] LIGI T,OOPKAUP K,TRUU M,et al. Characterization of bacterial communities in soil and sediment of a created riverine wetland complex using high-throughput 16S rRNA amplicon sequencing[J]. Ecological Engineering,2014,72:56 doi: 10.1016/j.ecoleng.2013.09.007
[67] THOMSON B C,OSTLE N J,MCNAMARA N P,et al. Effects of sieving,drying and rewetting upon soil bacterial community structure and respiration rates[J]. Journal of Microbiological Methods,2010,83(1):69 doi: 10.1016/j.mimet.2010.07.021
[68] PEPE-RANNEY C,KOECHLI C,POTRAFKA R,et al. Non-cyanobacterial diazotrophs mediate dinitrogen fixation in biological soil crusts during early crust formation[J]. The ISME Journal,2016,10(2):287 doi: 10.1038/ismej.2015.106
[69] DING L L,TIAN L L,LI J Y,et al. Grazing lowers soil multifunctionality but boosts soil microbial network complexity and stability in a subtropical grassland of China[J]. Frontiers in Microbiology,2023,13:1027097 doi: 10.3389/fmicb.2022.1027097
[70] SHENG Y Y,CONG W,YANG L S,et al. Forest soil fungal community elevational distribution pattern and their ecological assembly processes[J]. Frontiers in Microbiology,2019,10:2226 doi: 10.3389/fmicb.2019.02226
[71] WU X K,ZHANG G S,ZHANG W,et al. Variations in culturable bacterial communities and biochemical properties in the foreland of the retreating Tianshan No. 1 glacier[J]. Brazilian Journal of Microbiology,2018,49(3):443 doi: 10.1016/j.bjm.2018.01.001
[72] ALDEZABAL A,MORAGUES L,ODRIOZOLA I,et al. Impact of grazing abandonment on plant and soil microbial communities in an Atlantic Mountain grassland[J]. Applied Soil Ecology,2015,96:251 doi: 10.1016/j.apsoil.2015.08.013
[73] URBANOVÁ M,ŠNAJDR J,BALDRIAN P. Composition of fungal and bacterial communities in forest litter and soil is largely determined by dominant trees[J]. Soil Biology and Biochemistry,2015,84:53 doi: 10.1016/j.soilbio.2015.02.011
[74] LUO S,SCHMID B,DE DEYN G B,et al. Soil microbes promote complementarity effects among co-existing trees through soil nitrogen partitioning[J]. Functional Ecology,2018,32(7):1879 doi: 10.1111/1365-2435.13109
-
期刊类型引用(1)
1. 孙佳星,徐国荣,成鑫栋,王雨媛,吴江琪. 增温和放牧对河西盐渍化草地土壤理化性质及酶活性的影响. 水土保持学报. 2025(02): 309-317+347 .  百度学术
百度学术
其他类型引用(3)
-
其他相关附件
-
PDF格式
2023140附件 点击下载(624KB)
-




 下载:
下载: